raport3 v1 (PDF)
File information
Author: Behoston
This PDF 1.4 document has been generated by Writer / LibreOffice 5.0, and has been sent on pdf-archive.com on 21/02/2016 at 21:24, from IP address 93.105.x.x.
The current document download page has been viewed 570 times.
File size: 2.38 MB (7 pages).
Privacy: public file



File preview
Raport III
Mateusz Legięcki
nr indeksu 3444978
wydział MIM UW
Cele ćwiczenia
1. Zapoznanie się z komercyjnym programem do modelowania molekularnego – moe
2. Przeprowadzenie analizy dokowania ligandu
Wstęp
Analizowanym przez nas białkiem było 1OPL w układzie z ligandem P16 (dla obu
łańcuchów) oraz Myr (dla łańcucha A). Jest to receptor wzrostu naskórka. Jest to ludzkie białko
zaklasyfikowane jako transferaza. Długość białka wynosi 537. W pliku jest natomiast N-terminalna
część białka zawierająca 531 residuów. Mutacje występujące to: K29R, E30D, D382N. Ligand P16
w tym przypadku jest inhibitorem białka. W pliku znajdują się dwa łańcuchy, my analizujemy tylko
łańcuch A. Struktura białka jest pełna w okolicy miejsca wiązania ligandu. Struktura została
uzyskana poprzez eksperyment X-RAY i ma rozdzielczość 3.42Å. Wiemy, że białko łączy się z
ATP, oraz, że w miejsce wiązania mogą wchodzić inhibitory. Według bazy PFAM białko posiada
trzy domeny: SH2, SH3_1 oraz Pkinase_Tyr
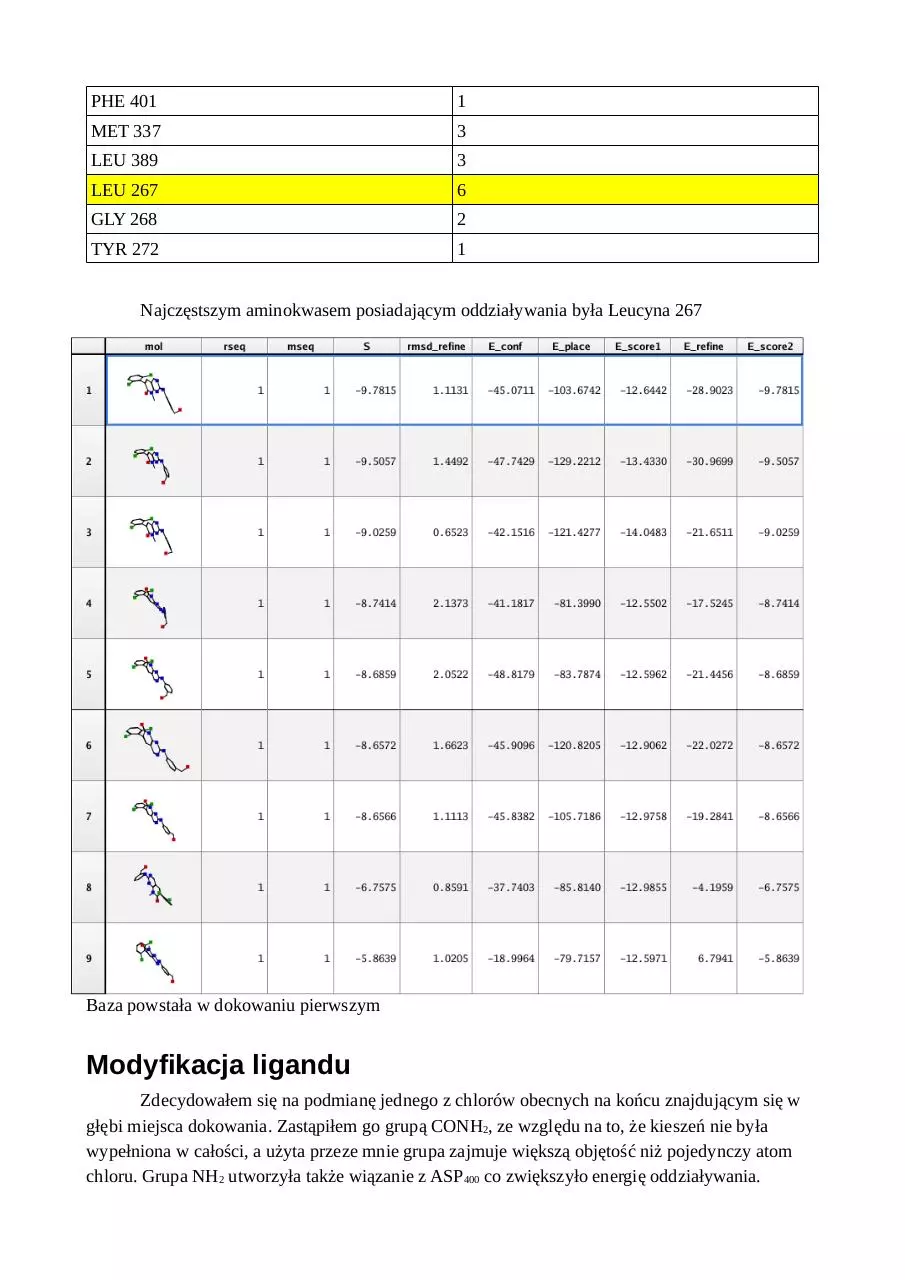
Dokowanie I
Pierwsze dokowanie jest dokowaniem
kontrolnym, dokujemy ten sam ligand do białka,
aby sprawdzić czy zadokuje się poprawnie w tej
samej pozycji. Służy nam to do oceny czy program
działa poprawnie dla naszego białka, nie jest to
idealny test, ale daje nam jakiś pogląd na jakość
wyników.
Parametry dokowania przedstawiono na
obrazku obok →
Analiza dokowania pierwszego
Ligand został zadokowany w poprawny sposób, co oznacza, że parametry domyślne są dla
nas wystarczająco dobre. Tylko mod nr 8 został zadokowany odwrócony, co nie spełniało naszych
założeń biologicznych, więc nie został wzięty pod uwagę w dalszej analizie.
Mody podzielono na następujące klastry:
Zawartość klastra
Średnia energia wewnątrz klastra
1,3
-6.7
6,7
-4.05
2
-5.1
5
-2.6
4
-4
9
-2.4
Jako najlepszy mod został wybrany (przez program – funkcja oceniająca oraz ta sama
konformacja co ligand przed dokowaniem) mod nr 2. Został on użyty do dalszych etapów, mimo
tego, że klaster nie ma najniższej energii.
Oryginalny ligand zaznaczony na kolor pomarańczowy, szarym kolorem oznaczono mod nr 2
Następujące aminokwasy brały udział w oddziaływaniach:
Aminokwas
Liczba wystąpień
ASP 344
3
ASN 341
1
VAL 275
1
PHE 401
1
MET 337
3
LEU 389
3
LEU 267
6
GLY 268
2
TYR 272
1
Najczęstszym aminokwasem posiadającym oddziaływania była Leucyna 267
Baza powstała w dokowaniu pierwszym
Modyfikacja ligandu
Zdecydowałem się na podmianę jednego z chlorów obecnych na końcu znajdującym się w
głębi miejsca dokowania. Zastąpiłem go grupą CONH2, ze względu na to, że kieszeń nie była
wypełniona w całości, a użyta przeze mnie grupa zajmuje większą objętość niż pojedynczy atom
chloru. Grupa NH2 utworzyła także wiązanie z ASP400 co zwiększyło energię oddziaływania.
Podstawienie ograniczyło także możliwości obrotu pierścienia względem reszty ligandu, ale nie w
100%.
Oddziaływania ligandu przed modyfikacją
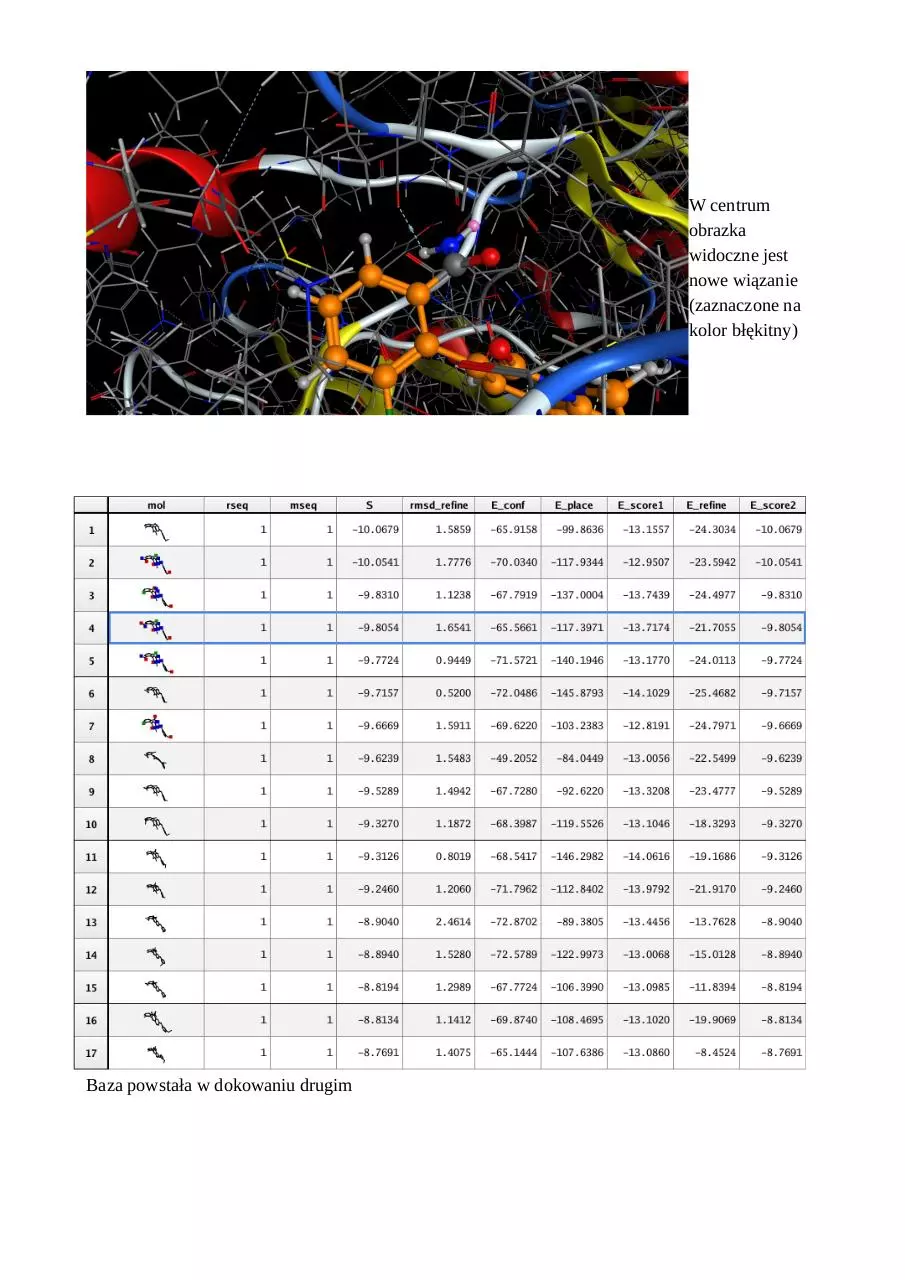
Oddziaływania ligandu po modyfikacji
W centrum
obrazka
widoczne jest
nowe wiązanie
(zaznaczone na
kolor błękitny)
Baza powstała w dokowaniu drugim
Dokowanie II
Użyto tych samych parametrów co w dokowaniu pierwszym.
Analiza dokowania drugiego
Wygenerowane mody zostały przeze mnie podzielone na klastry w następujący sposób:
Zawartość klastra
Średnia energia wiązań w klastrze
1,3,7,11
-8.05
2,4,5,10
-7.175
6,9
-5.5
8
-4
12
-7.4
13,15
0
14
-2.9
16
-6.4
17
-1.9
Następujące aminokwasy brały udział w oddziaływaniach:
Aminokwas
Liczba wystąpień
ASP 344
1
LYS 290
1
THR 338
1
ASN 341
4
THR 334
1
MET 337
11
LEU 389
11
ASP 400
5
LEU 267
7
GLY 268
1
TYR 272
1
Metionina 337 oraz Leucyna 389 są najbardziej istotnymi aminokwasami przy wiązaniu
tego ligandu.
Porównanie dokowania I i II
W dokowaniu pierwszym najczęściej oddziałującym aminokwasem była Leucyna 267 zaś w
drugim dokowaniu przeważały inne aminokwasy. Leucyna 267 wciąż występowała dość często
(siedmiokrotnie w porównaniu do jedenastu wystąpień dwóch innych aminokwasów).
Najniższa średnia energia klastra w dokowaniu drugim została obniżona – z -6.7 do -8.05.
Obniżona została też średnia energia ze wszystkich modów ogółem
z dokowania pierwszego = -4.45
z dokowania drugiego = -5.55882352941
Jednak wzrosła wariancja i w drugim dokowaniu występują klastry o energii wyższej niż najwyższa
energia klastra w dokowaniu pierwszym.
Z uwagi na modyfikacje, początkowe założenia biologiczne z dokowania pierwszego nie
były użyteczne w dokowaniu drugim
Wnioski
Modyfikacja ligandu zmniejszyła energię, lecz dała też o wiele więcej różnych klastrów co
znacznie zwiększa możliwości dalszych badań. Najczęściej oddziałujący aminokwas po dokowaniu
drugim wciąż miał wysoką frekwencję, jednak udało się uzyskać dwa aminokwasy częściej
tworzące oddziaływania.
Podsumowanie
Modyfikowanie ligandów może pomóc w zwiększeniu powinowactwa leku do celu, dzięki
nowym oddziaływaniom, bądź poprawie już istniejących.
Download raport3 v1
raport3_v1.pdf (PDF, 2.38 MB)
Download PDF
Share this file on social networks
Link to this page
Permanent link
Use the permanent link to the download page to share your document on Facebook, Twitter, LinkedIn, or directly with a contact by e-Mail, Messenger, Whatsapp, Line..
Short link
Use the short link to share your document on Twitter or by text message (SMS)
HTML Code
Copy the following HTML code to share your document on a Website or Blog
QR Code to this page

This file has been shared publicly by a user of PDF Archive.
Document ID: 0000341992.